-検索条件
-検索結果
検索 (著者・登録者: yang & b)の結果4,167件中、1から50件目までを表示しています

EMDB-37727: 
Cryo-ET structure of RuBisCO from 3.9 angstroms Synechococcus elongatus PCC 7942
手法: サブトモグラム平均 / : Kong WW, Jiang YL, Zhou CZ

EMDB-37728: 
Cryo-ET map of RuBisCO at 4.4 angstroms from Synechococcus elongatus PCC 7942 beta-carboxysome
手法: サブトモグラム平均 / : Kong WW, Jiang YL, Zhou CZ

EMDB-37729: 
Cryo-ET map of RuBisCO-SSUL at 5.9 angstroms from Synechococcus elongatus PCC 7942 beta-carboxysome
手法: サブトモグラム平均 / : Kong WW, Jiang YL, Zhou CZ

EMDB-37730: 
Cryo-ET map of RuBisCO at the outermost layer that is loosely attached to the shell of Synechococcus elongatus PCC 7942 beta-carboxysome
手法: サブトモグラム平均 / : Kong WW, Jiang YL, Zhou CZ

EMDB-37731: 
Cryo-ET map of RuBisCO at the outermost layer that is tightly attached to the shell of Synechococcus elongatus PCC 7942 beta-carboxysome
手法: サブトモグラム平均 / : Kong WW, Jiang YL, Zhou CZ

EMDB-39916: 
SARS-CoV-2 Omicron BA.1 spike trimer (x2-4P) in complex with 3 D1F6 Fabs (0 RBD up)
手法: 単粒子 / : Liu B, Gao X, Li Z, Chen Q, He J, Xiong X

EMDB-39917: 
SARS-CoV-2 Omicron BA.2 spike trimer (6P) in complex with 3 D1F6 Fabs (1 RBD up)
手法: 単粒子 / : Liu B, Gao X, Li Z, Chen Q, He J, Xiong X

EMDB-39918: 
SARS-CoV-2 Omicron BA.2 spike trimer (6P) in complex with 3 D1F6 Fabs (2 RBD up)
手法: 単粒子 / : Liu B, Gao X, Li Z, Chen Q, He J, Xiong X

EMDB-39919: 
SARS-CoV-2 Omicron BA.2 spike trimer (6P) in complex with D1F6 Fab, focused refinement of RBD region
手法: 単粒子 / : Liu B, Gao X, Li Z, Chen Q, He J, Xiong X

EMDB-39921: 
SARS-CoV-2 Omicron BA.4 spike trimer (6P) in complex with 3 D1F6 Fabs (1 RBD up)
手法: 単粒子 / : Liu B, Gao X, Li Z, Chen Q, He J, Xiong X

EMDB-39922: 
SARS-CoV-2 Omicron BA.4 spike trimer (6P) in complex with 3 D1F6 Fabs (2 RBD up)
手法: 単粒子 / : Liu B, Gao X, Li Z, Chen Q, He J, Xiong X

EMDB-39923: 
SARS-CoV-2 Omicron BA.4 spike trimer (6P) in complex with D1F6 Fab, focused refinement of RBD region
手法: 単粒子 / : Liu B, Gao X, Li Z, Chen Q, He J, Xiong X

EMDB-36068: 
Monkeypox virus DNA replication holoenzyme F8, A22 and E4 in complex with a DNA duplex and dCTP
手法: 単粒子 / : Xu Y, Wu Y, Wu X, Zhang Y, Yang Y, Li D, Yang B, Gao K, Zhang Z, Dong C, Tang X, Dong H

EMDB-36069: 
Monkeypox virus DNA replication holoenzyme F8, A22 and E4 in complex with a DNA duplex and cidofovir diphosphate
手法: 単粒子 / : Xu Y, Wu Y, Wu X, Zhang Y, Yang Y, Li D, Yang B, Gao K, Zhang Z, Dong C, Tang X, Dong H

PDB-8j8f: 
Monkeypox virus DNA replication holoenzyme F8, A22 and E4 in complex with a DNA duplex and dCTP
手法: 単粒子 / : Xu Y, Wu Y, Wu X, Zhang Y, Yang Y, Li D, Yang B, Gao K, Zhang Z, Dong C

PDB-8j8g: 
Monkeypox virus DNA replication holoenzyme F8, A22 and E4 in complex with a DNA duplex and cidofovir diphosphate
手法: 単粒子 / : Xu Y, Wu Y, Wu X, Zhang Y, Yang Y, Li D, Yang B, Gao K, Zhang Z, Dong C

EMDB-36061: 
Monkeypox virus DNA replication holoenzyme F8, A22 and E4 complex in a DNA binding form
手法: 単粒子 / : Xu Y, Wu Y, Wu X, Zhang Y, Yang Y, Li D, Yang B, Gao K, Zhang Z, Dong C, Tang X, Dong H

PDB-8j86: 
Monkeypox virus DNA replication holoenzyme F8, A22 and E4 complex in a DNA binding form
手法: 単粒子 / : Xu Y, Wu Y, Wu X, Zhang Y, Yang Y, Li D, Yang B, Gao K, Zhang Z, Dong C

EMDB-42949: 
Microtubule inner proteins in the 48-nm doublet microtubule from the proximal region of Tetrahymena thermophila strain K40R
手法: 単粒子 / : Legal T, Bui KH, Yang SK

EMDB-43931: 
CryoEM structure of activated CRAF/MEK/14-3-3 complex with NST-628
手法: 単粒子 / : Quade B, Cohen SE, Huang X

EMDB-43932: 
Activated CRAF/MEK heterotetramer from focused refinement of CRAF/MEK/14-3-3 complex
手法: 単粒子 / : Quade B, Cohen SE, Huang X

PDB-9axa: 
CryoEM structure of activated CRAF/MEK/14-3-3 complex with NST-628
手法: 単粒子 / : Quade B, Cohen SE, Huang X

PDB-9axc: 
Activated CRAF/MEK heterotetramer from focused refinement of CRAF/MEK/14-3-3 complex
手法: 単粒子 / : Quade B, Cohen SE, Huang X

EMDB-43705: 
HIV-1 wild-type intasome core
手法: 単粒子 / : Li M, Craigie R

EMDB-43756: 
HIV-1 P5-IN intasome core
手法: 単粒子 / : Li M, Craigie R
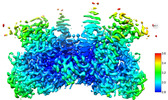
EMDB-43761: 
HIV-1 intasome core assembled with wild-type integrase, 1F
手法: 単粒子 / : Li M, Craigie R
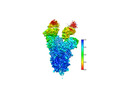
EMDB-37240: 
SARS-CoV-2 Omicron spike in complex with 5817 Fab
手法: 単粒子 / : Cao L, Wang X

EMDB-37241: 
The interface structure of Omicron RBD binding to 5817 Fab
手法: 単粒子 / : Cao L, Wang X

EMDB-38200: 
Cryo-EM structure of OSCA1.2-liposome-inside-in open state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38503: 
Cryo-EM structure of OSCA1.2-liposome-inside-out closed state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38611: 
Cryo-EM structure of OSCA3.1-1.1ver(Y367N-G454S-Y458I)-open/open state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38612: 
Cryo-EM structure of OSCA3.1-1.1ver(Y367N-G454S-Y458I)-open/'desensitized' state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38614: 
Cryo-EM structure of OSCA1.2-DOPC-1:20-contracted1 state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38615: 
Cryo-EM structure of OSCA1.2-DOPC-1:20-contracted2 state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38721: 
Cryo-EM structure of OSCA1.2-DOPC-1:20-expanded state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38722: 
Cryo-EM structure of OSCA1.2-DOPC-1:50-betaCD state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38723: 
Cryo-EM structure of OSCA3.1-2E(R611E-R619E)-closed/open state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38724: 
Cryo-EM structure of OSCA3.1-2E(R611E-R619E)-closed/'desensitized' state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38725: 
Cryo-EM structure of OSCA3.1-GDN state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38726: 
Cryo-EM structure of OSCA3.1-liposome-inside-in state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38727: 
Cryo-EM structure of OSCA1.2-V335W-DDM state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38728: 
Cryo-EM structure of OSCA1.2-DOPC-1:50-contracted state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38729: 
Cryo-EM structure of OSCA1.2-DOPC-1:50-expanded state
手法: 単粒子 / : Zhang Y, Han Y

EMDB-38730: 
Cryo-EM structure of TMEM63B-Digitonin state
手法: 単粒子 / : Zhang Y, Han Y
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について









 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
